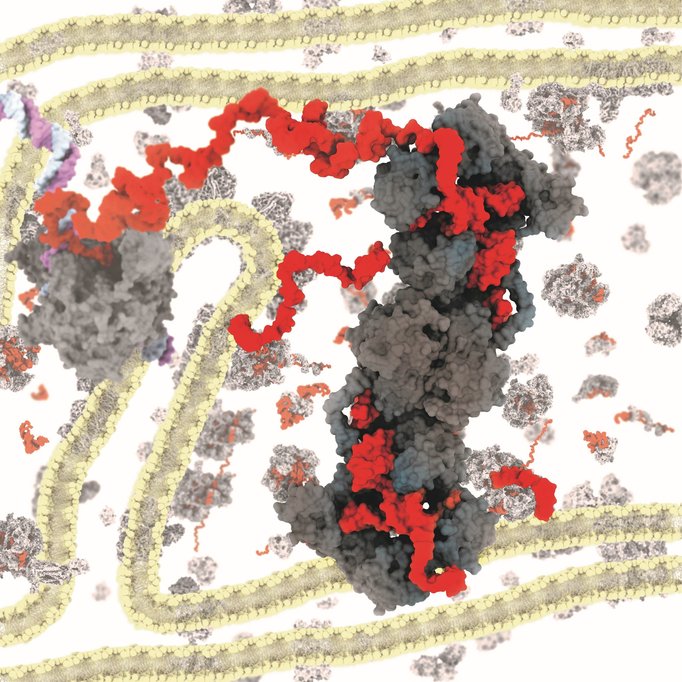
Mitochondrien sind kleine Organellen innerhalb menschlicher Zellen, die die Energie im Körper bereitstellen. Um das zu ermöglichen, besitzen sie ein eigenes Genom und spezielle Gene, die aktiviert werden müssen. Ein wichtiger Schritt in der mitochondrialen Genaktivierung ist die sogenannte RNA-Prozessierung, bei der Vorläufermoleküle in funktionale RNA Abschnitte zerteilt werden. Störungen dieses Prozesses, z. B. durch genetische Defekte, führen zu schweren Krankheiten. Der molekulare Mechanismus der mitochondrialen RNA-Prozessierung ist jedoch weitgehend unbekannt. Forschende am Max-Planck-Institut für biophysikalische Chemie (MPI-BPC) und an der Universitätsmedizin Göttingen (UMG) konnten nun im Detail den ersten Schritt des Mechanismus aufklären. Sie nutzten hochauflösende Kryo-Elektronenmikroskopie um die dreidimensionale Struktur einer mitochondrialen RNA-Schneide-Maschinerie, der sogenannten Ribonuklease P (mtRNase P), zu bestimmen. Dadurch erlangten die Forschenden erstmals detaillierte Einblicke in den molekularen Mechanismus der mtRNase P. Die Ergebnisse liefern nicht nur wichtige Erkenntnisse für die Grundlagenforschung, sondern erklären auch, weshalb bestimmte Mutationen in den Protein-Untereinheiten der mtRNase P zu Krankheiten führen können.
Menschliche Zellen enthalten zwei Genome. Zusätzlich zum Erbgut im Zellkern existiert ein zweiter Satz genetischen Materials in den Mitochondrien. Letzterer ist ein Überbleibsel ihrer evolutionären Vorfahren. Denn Mitochondrien haben sich vermutlich vor Millionen von Jahren aus bestimmten Bakterien gebildet, die von den Vorfahren unserer heutigen Zellen aufgenommen wurden. Heute leisten die Mitochondrien einen lebenswichtigen Dienst, denn sie produzieren mithilfe von Sauerstoff den chemischen Energieträger Adenosin-Triphosphat (ATP). Um dies leisten zu können, müssen die mitochondrialen Gene aktiviert werden.
Der erste Schritt der Genaktivierung besteht aus der Transkription, bei der die in der DNA gespeicherte genetische Information in RNA kopiert wird. Im Gegensatz zum Erbgut im Zellkern wird das mitochondriale Erbgut nicht Gen für Gen transkribiert. Stattdessen wird fast das gesamte mitochondriale Genom auf einen Schlag in eine lange RNA überschrieben. Diese enthält dann verschiedene Abschnitte, in denen unterschiedliche Arten von genetischen Informationen enthalten sind. Um ihrer jeweiligen Funktion nachzukommen, müssen diese Abschnitte getrennt werden – ein Prozess, den man als RNA-Prozessierung bezeichnet.
In Mitochondrien ist eine spezialisierte RNA-Schneide-Maschinerie für den ersten Schritt dieses Prozesses verantwortlich, die mitochondriale Ribonuklease P (mtRNase P). Sie erkennt bestimmte genetische Abschnitte der RNA, die sogenannten Transfer-RNAs (tRNAs), und zerschneidet sie präzise an deren Anfang. Zusätzlich verändert mtRNase P auch die chemische Struktur der RNA an einer ganz bestimmten Stelle durch Methylierung. Darauf folgen mehrere weitere Prozessierungsschritte, bevor am Ende die verschiedenen RNA-Typen ihre Funktion in den Mitochondrien übernehmen können. Funktioniert die RNA-Prozessierung nicht ordnungsgemäß, führt das zur Fehlfunktion der Mitochondrien und infolgedessen zu schwerwiegenden Erkrankungen, sogenannten Mitochondriopathien. Der genaue molekulare Mechanismus der mitochondrialen RNA-Prozessierung durch mtRNase P war allerdings bisher unbekannt, da sich die mitochondriale RNA-Schneidemaschine grundlegend unterscheidet von den Enzymen, die ähnliche Aufgaben im Zellkern oder in Bakterien ausführen.
Erste hochaufgelöste Struktur der menschlichen mitochondrialen RNA-Schnittmaschine
Forschenden am MPI-BPC sowie an der UMG ist es nun erstmals gelungen, die genaue Funktionsweise der mitochondrialen RNA-Schneidemaschine zu entschlüsseln. Dazu nutzten die Wissenschaftler um Gruppenleiter und Juniorprofessor Hauke Hillen die Kryo-Elektronenmikroskopie, um die dreidimensionale Struktur der mtRNase P zu bestimmen. Die hochaufgelöste Struktur ermöglicht den Forschenden, die Abläufe bei der mitochondrialen RNA-Prozessierung erstmals in atomarem Detail zu bestimmen. Sie zeigt, wie die verschiedenen Protein-Untereinheiten miteinander interagieren, um die charakteristische Form der tRNA zu erkennen und binden zu können. Erstaunlicherweise interagiert die mtRNase P dabei mit einem vollkommen anderen Bereich der tRNA als alle bisher bekannten RNA-Prozessierungsenzyme. „Das war zunächst sehr überraschend!“, sagt Hillen. „Doch es ergibt Sinn, denn mitochondriale tRNAs unterscheiden sich stark von ‘normalen‘ tRNAs, insbesondere in den Bereichen, die von anderen Prozessierungsenzymen gebunden werden. Um dennoch eine Bindung zu ermöglichen, interagiert mtRNase P zusätzlich mit einem anderen, stark konservierten Bereich der tRNA.“, erklärt er weiter. Dies ermöglicht es der molekularen Schneidemaschine, die RNA exakt so zu binden, dass sie an der richtigen Stelle zerschnitten und methyliert wird. „Dies könnte auch erklären, warum sich in den Mitochondrien von Säugetieren eine solch hoch spezialisierte RNA-Prozessierungsmaschine entwickelt hat. Denn die ungewöhnliche Art der tRNA-Bindung wird durch Proteinuntereinheiten vermittelt, die es nur in der mitochondrialen RNase P, nicht aber in verwandten Enzymen im Zellkern oder in Bakterien gibt“, erläutert Arjun Bhatta, Doktorand in Hillens Gruppe und Erstautor der Studie.
Molekulare Einblicke in die Entstehung mitochondrialer Erkrankungen
Neben grundlegenden Einblicken in die Abläufe der RNA-Prozessierung in Mitochondrien, liefert die Struktur der mtRNase P auch neue Erkenntnisse zur Ursache bestimmter mitochondrialer Erkrankungen. Bereits zuvor wurde gezeigt, dass Mutationen in den Protein-Untereinheiten der mtRNase P zu schweren Krankheiten auslösen können. Anhand der Struktur konnten die Forscher nun ermitteln, wo genau sich diese Veränderungen befinden und so Vorhersagen über deren Auswirkung auf die Funktion des Enzyms machen. Dabei stellte sich heraus, dass einige krankheitsverursachende Mutationen in den Teilen der mtRNase P auftreten, die direkt an der RNA-Bindung beteiligt sind und so vermutlich die Funktion des Enzyms beeinträchtigen. Somit liefert die Struktur eine mögliche molekulare Erklärung für die durch Mutationen ausgelösten mitochondrialen Erkrankungen.
In Zukunft wollen Hillen und sein Team auf diesen ersten Ergebnissen aufbauen und die weiteren Schritte der mitochondrialen RNA-Prozessierung im Detail untersuchen. (hh)
Kontakt
Prof. Dr. Hauke Hillen
Forschungsgruppe Struktur und Funktion molekularer Maschinen
+49 551 201-2884
hauke.hillen@...
Publikation
Bhatta A, Dienemann C, Cramer P & Hillen HS
Structural basis of RNA processing by human mitochondrial RNase P
Nat Struct Mol Biol (2021)
Source