Damit Viren sich vermehren können, benötigen sie zumeist die Unterstützung der von ihnen befallenen Zellen. Nur im Zellkern ihrer Wirtszellen finden sie die Maschinen, Proteine und Bausteine, mit denen sie ihr genetisches Material vervielfachen können, bevor sie weitere Zellen infizieren.
Doch nicht alle Viren finden den Weg in den Zellkern. Einige verbleiben außerhalb im sogenannten Zytoplasma und müssen aus eigener Kraft ihr Erbgut verdoppeln. Den dafür notwendigen „Maschinenpark“ bringen sie selbst mit. Eine wesentliche Rolle übernimmt dabei eine spezielle Nanomaschine, kombiniert mit diversen Untereinheiten: die RNA-Polymerase. Diese zelluläre Kopiermaschine liest die genetische Information vom Erbgut des Virus ab und übersetzt sie in die Boten-RNA – ein langes Molekül, das als Blaupause für die im Erbgut kodierten Proteine dient. Dieser Vorgang heißt Transkription.
Wissenschaftlern um Patrick Cramer, Direktor und Leiter der Abteilung Molekularbiologie am Max-Planck-Institut (MPI) für biophysikalische Chemie, und Utz Fischer von der Julius-Maximilians-Universität (JMU) Würzburg ist es jetzt erstmals gelungen, die Struktur dieser Nanomaschinen aus Pockenviren dreidimensional und in atomarer Auflösung darzustellen. Sie arbeiteten dabei mit Vaccinia, einem DNA-Virus. Dieser für den Menschen völlig harmlose Erreger ist nur Grundlage aller Impfstoffe gegen Pockenerkrankungen. Er wird auch in der sogenannten onkolytischen Virotherapie erprobt, um Krebserkrankungen zu bekämpfen.
Eine molekulare Klammer, die alles zusammenhält
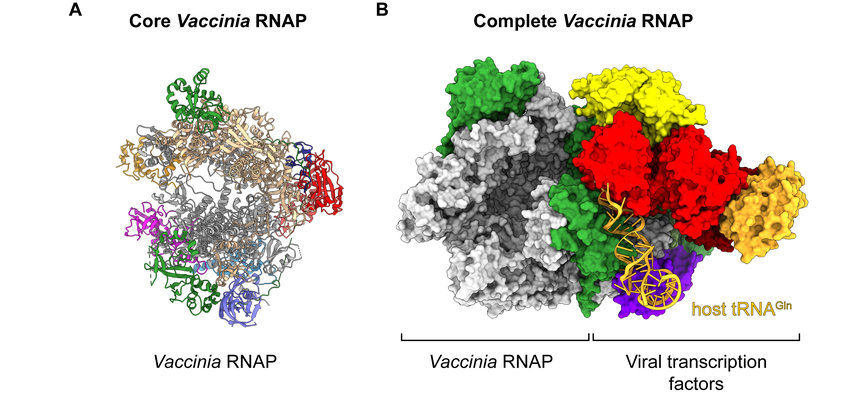
„Die RNA-Polymerase des Vaccinia-Virus existiert im Wesentlichen in zwei Erscheinungsformen: dem eigentlichen Kernenzym und einem noch größeren Komplex, der dank hinzugefügter Untereinheiten über weitere, spezielle Funktionalitäten verfügt“, erklärt Fischer. Das Kernenzym gleicht der molekularen Kopiermaschine, die natürlicherweise in lebenden Zellen vorkommt und intensiv von der Abteilung Cramer erforscht wird: der RNA-Polymerase II. Der zweite Komplex der Vaccinia-RNA-Polymerase ist ein Alleskönner. Er besteht aus zahlreichen Untereinheiten und führt den gesamten Transkriptionsprozess für den Virus aus. So kann der Virus sich vermehren.
Zusammengehalten wird der Komplex von einem Molekül, welches das Virus aus seiner Wirtszelle entwendet: einer sogenannten Transfer-RNA (tRNA). Diese Art von Molekülen spielt normalerweise keine Rolle in der Transkription, sondern liefert die Aminosäure-Bausteine für die Proteinherstellung. Wäre die Wirts-tRNA nicht beteiligt, fiele der riesige Komplex wahrscheinlich auseinander.
Um der Funktionsweise der viralen RNA-Polymerase auf die Spur zu kommen, ermittelten die Forscher – unter Beteiligung von Henning Urlaub, Forschungsgruppenleiter am MPI für biophysikalische Chemie – ihre dreidimensionale Struktur während unterschiedlichen Schritten der Transkription. „Mit den neuen Erkenntnissen können wir nun den gesamten Prozess der Viren-Vermehrung nachvollziehen. Wie in einem Film lässt sich verfolgen, wie diese Nanomaschine auf atomarer Ebene funktioniert und wie die einzelnen Abläufe choreografiert sind“, sagt Cramer. Sein Mitarbeiter, Strukturbiologe Hauke Hillen, fügt hinzu: „Besonders erstaunlich ist, wie sich die Bausteine der Maschine nach dem Start der Transkription neu anordnen, um die Synthese des RNA-Produkts voranzutreiben – dieser Komplex ist wirklich sehr dynamisch.“
Ein Supermikroskop liefert die nötigen Daten
Die entsprechenden Daten liefert ein Gerät, das die Strukturanalyse in den vergangenen Jahren revolutioniert hat – das Kryo-Elektronenmikroskop. Es ermöglicht Bilder mit einer Auflösung, die sich in der Größenordnung von Atomen bewegt. Solche Mikroskope nutzen neben dem MPI für biophysikalische Chemie auch immer mehr deutsche Universitäten, ermöglicht durch Fördergelder des Bundesministeriums für Bildung und Forschung und der Deutschen Forschungsgemeinschaft. Für ihre Aufnahmen haben die Forscher die RNA-Polymerase in unterschiedlichen Phasen der Transkription schockgefroren und „fotografiert“. Auf diese Weise entstanden Millionen Schnappschüsse der Nanomaschine während ihrer Arbeit, die die Forscher anschließend zu einem Gesamtbild zusammensetzten. Rund sechs Monate mussten Hillen und sein Würzburger Kollege Clemens Grimm mithilfe von Computern tüfteln, bis sie aus mehreren Terabyte Daten räumliche Modelle der Polymerase-Komplexe entwickelt hatten. Mit einer 3D-Brille kann sich nun jeder den Komplex ansehen, beliebig drehen und in seine Untereinheiten zerlegen.
Die neuen Erkenntnisse bieten unter anderem die Möglichkeit, auf den viralen Vermehrungszyklus Einfluss zu nehmen, was therapeutisches Potenzial mit sich bringt. Aktuell laufen weltweit Studien, die Vaccinia-Viren im Kampf gegen Krebs einsetzen. Dass speziell optimierte Vaccinia-Viren sogar Tumore verkleinern und kleinste Metastasen aufspüren können, hat bereits die Firma Genelux in Tierversuchen und an Patienten zeigen können.
Kontakt
Prof. Dr. Patrick Cramer
Direktor, Abteilung Molekularbiologie
+49 551 201-2800
patrick.cramer@...
Carmen Rotte
Leiterin der Presse- und Öffentlichkeitsarbeit
+49 551 201-1304
carmen.rotte@...
Originalpubikationen
Grimm C, Hillen HS, Bedenk H, Bartuli J, Neyer S, Zhang Q, Hüttenhofer A, Erlach M, Dienemann C, Schlosser A, Urlaub H, Böttcher B, Szalay A, Cramer P, Fischer U
Structural basis of poxvirus transcription: Vaccinia RNA polymerase complexes
Cell, December 12 (2019)
Hillen HS, Bartuli J, Grimm C, Dienemann C, Bedenk H, Szalay A, Fischer U, Cramer P
Structural basis of poxvirus transcription: transcribing and capping Vaccinia complexes
Cell, December 12 (2019)